En un avance significativo en la comprensión de los mecanismos moleculares de la meiosis, un estudio publicado en la revista Proceedings of the National Academy of Sciences (PNAS) abre nuevas vías para la investigación en el campo. Esta investigación, financiada por el Ministerio de Ciencia, Innovación y Universidades (MICIU) y la Junta de Castilla y León, tiene implicaciones importantes en la fertilidad y las enfermedades genéticas.
Las células humanas y animales son diploides, es decir, proceden de la fusión de dos gametos o células haploides que tienen 23 pares de cromosomas cada una. La producción de gametos es un proceso complejo, que requiere una forma altamente especializada de división celular llamada meiosis, explica Alberto M. Pendás, investigador del CIC y autor del estudio. Durante la meiosis, el número de cromosomas se reduce a la mitad, creando espermatozoides y óvulos a partir de células progenitoras diploides.
Los sobrecruzamientos entre cromosomas provocan uniones físicas entre pares de cromosomas homólogos (paterno y materno) que generan el intercambio de segmentos cromosómicos entre ambos cromosomas. Este proceso de recombinación y su posterior reparto aleatorio genera nuevas combinaciones de genes que son esenciales para mantener la diversidad genética de la progenie y de la población, siendo esencial para la evolución de las especies.
Es fundamental un control exhaustivo de los mecanismos implicados en este delicado proceso, porque los errores pueden dar lugar a células sexuales aneuploides (número anormal de cromosomas), abortos espontáneos, infertilidad o enfermedades genéticas, como el síndrome de Down, explica Elena Llano, profesora titular del departamento de Fisiología de la Universidad de Salamanca y autora del estudio.
El estudio describe una ruta molecular conservada evolutivamente desde las levaduras hasta los mamíferos, que es fundamental para comprender cómo se producen los sobrecruzamientos y cómo se configura el paisaje de la recombinación meiótica en eucariotas. En este sentido, se resalta la importancia evolutiva de las ligasas E3 en la meiosis. Las E3 ligasas son enzimas que unen pequeñas proteínas (SUMO o Ubiquitina) a otras proteínas más grandes, modulando su función, localización o estabilidad.
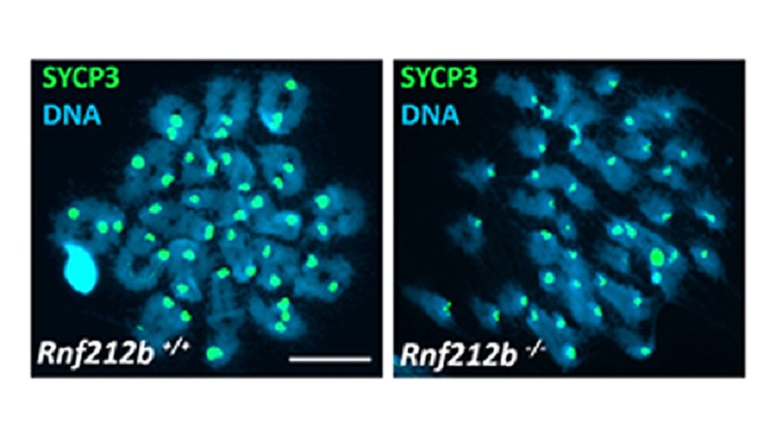
El grupo de investigación ha identificado que la E3 ligasa RNF212B es un factor crucial en la formación y maduración de los sobrecruzamientos. Su ausencia provoca que no haya sobrecruzamientos entre cromosomas homólogos, lo que impide que estos se repartan adecuadamente en los gametos. Los resultados explican la razón por la cual personas con defectos en el gen RNF212B sean estériles (azoospérmicos).
RNF212B asegura la maduración adecuada de los entrecruzamientos, para que los cromosomas homólogos se unan entre sí durante las primeras fases de la meiosis y eviten errores en el reparto de los cromosomas que provocan enfermedades reproductivas graves, destaca Pendás.
Este trabajo de investigación no solo mejora nuestra comprensión de la meiosis, sino que también subraya la complejidad de los procesos que aseguran la segregación precisa de los cromosomas y la generación de gametos viables, apunta Yazmine B. Condezo, coautora del estudio. Por ello, estas nuevas aportaciones pueden allanar el camino para establecer nuevas estrategias que permitan abordar los desafíos reproductivos y las enfermedades genéticas.
Comprender las funciones e interacciones de la ligasa E3 RNF212B podría llevar a enfoques terapéuticos novedosos para abordar los errores meióticos y mejorar la salud reproductiva, así como para el desarrollo de nuevos métodos anticonceptivos no hormonales y reversibles para los hombres. “La continua exploración de estos mecanismos promete avanzar nuestro conocimiento sobre la regulación genética y su impacto en la salud humana”, concluye Raquel Sainz-Urruela, coautora del estudio.
Referencia científica: Yazmine B. Condezo et al. RNF212B E3 ligase is essential for crossover designation and maturation during male and female meiosis in the mouse. Proceedings of the National Academy of Sciences (PNAS). DOI: 10.1073/pnas.2320995121